前几期,我们讲了单细胞转录组与普通转录组关联分析的优势、分析内容、应用思路。这两种转录组技术关联的核心优势如下:
1. 低投入,高性价比,助力研究者发表高分文章。
2. 少单细胞样本,多普通转录组样本的实验设计思路。
3. 两种转录组技术从双维度诠释细胞/基因表达机制。
4. 流式或LMD的普通转录组数据可以更准确的对单细胞数据做细胞注释或注释的结果验证。
5. 将普通转录组的实验效应表型数据映射到单细胞分辨率上进行核心细胞的机制研究。
6. 通过去卷积分析研究分化发育、肿瘤发展等领域,低成本的策略研究随时间发育的细胞比例和功能。
下面我通过两篇文献案例,对两种转录组关联的应用思路进行系统性的解读,希望可以帮助大家对两种转录组关联技术的应用有更深刻的理解,其中第一篇为基迪奥合作客户的项目文章。
案例一
分化发育研究(基迪奥客户文章)
中文题目:
单细胞转录组揭示苔藓类植物地钱的成熟和衰老轨迹[1]
英文题目:
The maturation and aging trajectory of Marchantia polymorpha at single-cell resolution
发表期刊:
Developmental Cell (IF=11.7994)
发表时间:
2023年8月
合作单位:
中国科学院分子植物科学卓越创新中心
研究目的
苔藓植物是植物界最原始的类群之一,它们生长在湿润和阴暗的环境中,没有根、茎和叶等器官,也不能通过维管束进行水分和养分的输送。尽管苔藓植物在进化上具有重要性,并且拥有相对简单的植株构造,但是人们对于其细胞类型组成以及成熟和衰老的轨迹还知之甚少。随着单细胞技术的发展,这一问题得以解决。该研究以苔类植物地钱(Marchantia polymorpha)为模式,利用时间分辨的单细胞转录组测序技术描绘了全球首张苔藓类植物的细胞图谱,研究了成熟和衰老的轨迹。
实验设计
(1)scRNA-seq:收集不同发育阶段(D0、D1、D3、D6、D11、D31)的休眠芽和发育中的菌体制备原生质体。
(2)bulk RNA-seq:将31日龄的菌体(D31)从尖端到基部切成6条,宽度为1 mm进行切片的bulk RNA-seq。同时采集不同发育阶段(D1 ~ D31)的菌体和休眠芽(D0)菌体约100 mg进行bulk RNA-seq。
研究结果
1.首张苔藓类植物的细胞图谱
将46136个细胞聚类为21个亚群,有趣的是,在UMAP图上,胚芽细胞(第0天的样本)与菌体细胞分离,表明在萌发期间(即从第0天到第1天),转录组水平上存在大量细胞重编程。相比之下,我们观察到从第1天到第31天,样品中的转录组逐渐发生变化(图1C和1D)。综上所述,作者通过不同发育阶段的scRNA-seq对苔藓菌体进行了全面的细胞普查。
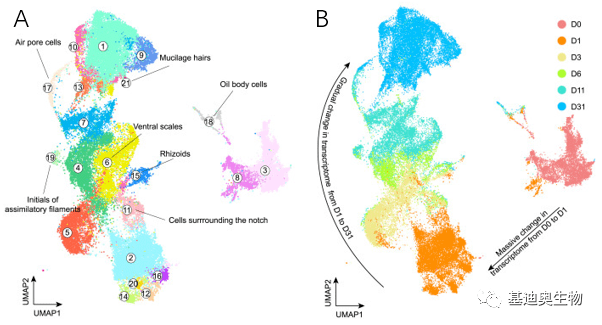
图1 苔藓类的细胞图谱
2.菌体的空间老化轴研究
同一时间点的菌体细胞在UMAP上倾向于聚集在一起,表明随着菌体的成熟和衰老,转录组逐渐发生变化。为了验证这一假设,作者使用了基于Pearson相关系数和回归模型的新开发的scRNA-seq算法Scissor来研究菌体的发育年龄。回归系数为非零的细胞分别被划分为剪刀阳性细胞(红点)和剪刀阴性细胞(蓝点),它们分别与成熟和衰老呈正相关和负相关,并且发现衰老相关基因在第31天的细胞中高度富集。
每个菌体都包含了不同发育时间的组织,从年轻的、活跃生长的尖端区域到较老的、成熟的基底部分,这表明scRNA-seq显示的观察到的渐进式衰老实际上可能反映了菌体中尖端到基底的衰老轴。为了证实这一假设,作者对菌体进行切割组织的bulk RNA-seq,将31天大的菌体从头到尾切成6片。将得到的切片组织RNA-seq数据与不同时间点的整体组织RNA-seq和伪单细胞RNA-seq进行相关性比较,结果发现切片1-3、4和5分别与第1天、第3-11天和第31天的整体和伪整体样品对齐。这些结果共同表明菌体中脉有一个渐进的老化轴(即空间老化轴)。
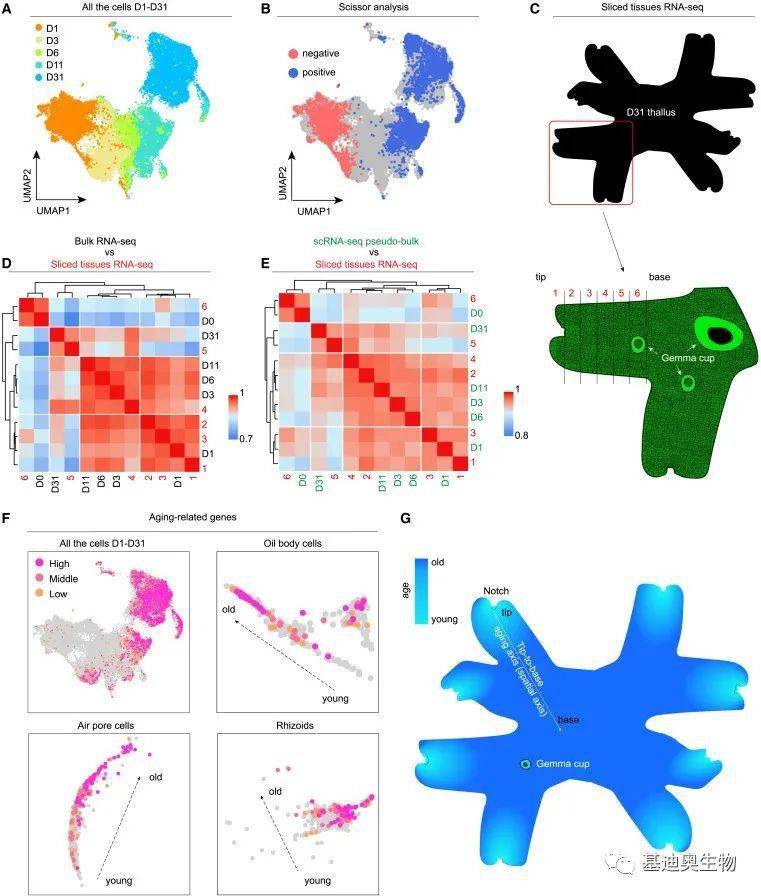
图2 沿中脉的尖端到基部的成熟和老化轴
3.油体细胞的成熟和衰老功能
接着作者在单细胞分辨率下研究了菌体特化细胞类型(油体细胞)的成熟和衰老。对油体细胞进行再分群分析,油体子亚群老油体O2富含衰老和光合作用相关的基因。这些结果表明,靠近中脉顶端分生组织区域的幼年期油细胞主要进行次生代谢化合物的合成工作,这些有毒代谢物的积累是地钱抵御昆虫取食的主要方式。随着时间的推移,叶状体基部的成年期油细胞则逐渐转向进行光合作用,提供叶状体发育所需的能量。
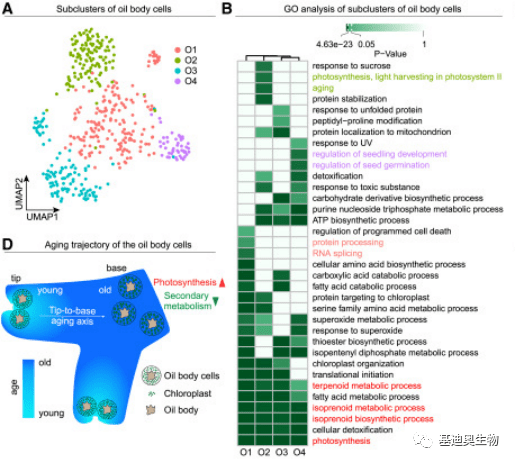
图3 油体再分群分析
研究结论
本研究绘制了全球首张苔藓类植物的细胞图谱。该图谱揭示了地钱由休眠孢芽(dormant gemma)发育成为成熟叶状体(mature thallus)的细胞类型动态变化轨迹,并在单细胞分辨率下确定了叶状体的两个成熟和衰老轨迹:即组织和器官沿中脉顶端(tip)至基部(base)逐渐成熟衰老的空间衰老轴(spatial axis)以及顶端分生组织活性随时间逐渐下降的时间衰老轴(temporal axis)。
案例二
肿瘤免疫浸润
中文题目:
单细胞转录组研究乳腺癌三级淋巴结构和B细胞的肿瘤浸润[2]
英文题目:
Single-cell tranome sequencing of B-cell heterogeneity and tertiary lymphoid structure predicts breast cancer prognosis and neoadjuvant therapy efficacy
发表期刊:
Clin Transl Med(IF=10.6002)
发表时间:
2023年8月
研究目的
乳腺癌(BC)是一种高度异质性的恶性肿瘤。B细胞,尤其是聚集在三级淋巴结构(TLS)中的B细胞,在抗肿瘤免疫中起着重要作用。然而,单细胞分辨率下的B细胞异质性及其在骨髓细胞TLS中的临床意义需要进一步探讨。
实验设计
(1)scRNA-seq:从14例BC患者中取原发肿瘤病变及周围正常组织进行单细胞转录组测序,质控后获得124587个高质量细胞。
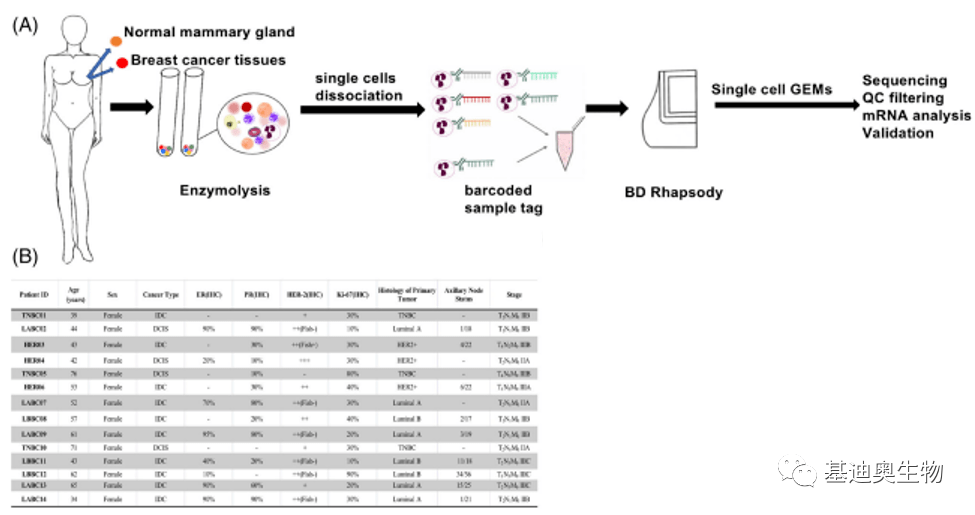
(2)bulk RNA-seq:公共数据库中BC队列(GSE7390, GSE58812)数据。
研究结果
1.单细胞转录组识别乳腺癌的肿瘤微环境
对单细胞数据进行无监督聚类,鉴定出7个细胞类型,细胞频率分析发现这些细胞类型在患者中分布不均匀,证明了人类BC的极端异质性。
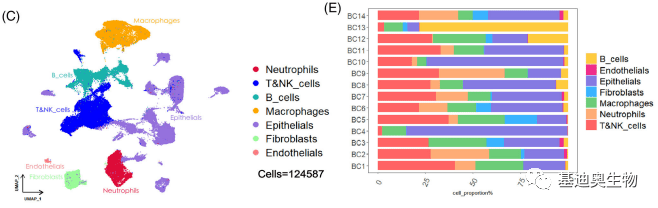
图1 BC细胞图谱
2.乳腺癌中B细胞浸润性肿瘤和发展过程
对B细胞进行再分群分析,获得5个B细胞亚群,这些亚群在个体中可以观察到显著的差异,说明了B细胞浸润肿瘤的重要性。通过对B细胞亚群进行拟时分析,红色和绿色箭头实现了细胞发育的两条时间轨迹。在每个亚群中发现了随时间变化较大的差异基因,其中CXCR4和STMN1可以增强免疫功能和促进肿瘤扩散的基因,从最初的高表达到最终的下降同时逐渐下降,说明了B细胞抗肿瘤和肿瘤能力的矛盾。
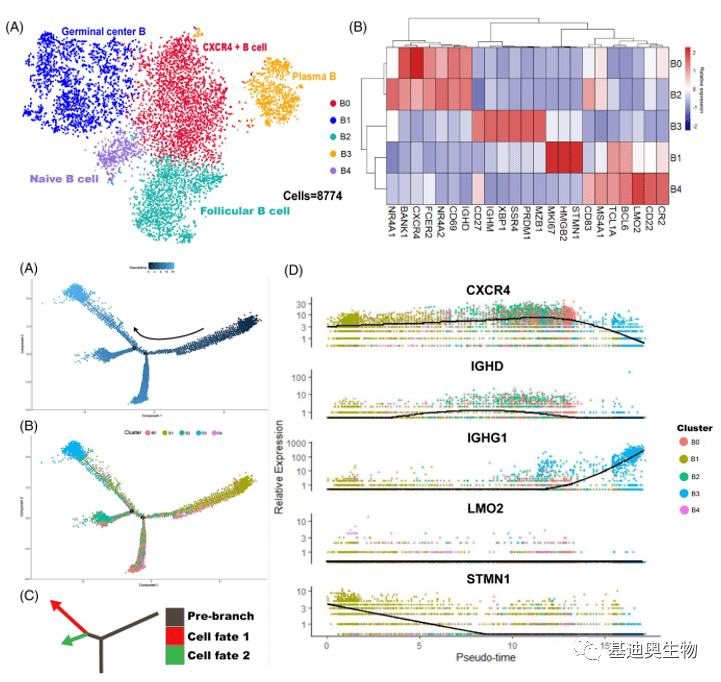
图2 B细胞再分群和拟时分析
3.肿瘤微环境之间的细胞通讯网络
利用CellphoneDB,阐明了成纤维细胞和多种免疫细胞类型中配体和受体之间的相互作用。分析的重点是9例乳腺癌(BC)患者的三级淋巴样结构(TLS)阴性(左)和TLS阳性(右)队列。TLS阳性细胞表达相对较低水平的FAS-FASLG受体-配体对,这与免疫耐受有关。免疫检查点相关受体配体对CTLA4-CD86、CTLA4-CD80、LGALS9-HAVCR2、CD80-CD274和LAGALS9-CD44在TLS阴性细胞中的含量相对高于TLS阳性细胞。这些发现指出了TLS阴性患者中潜在的免疫抑制微环境。
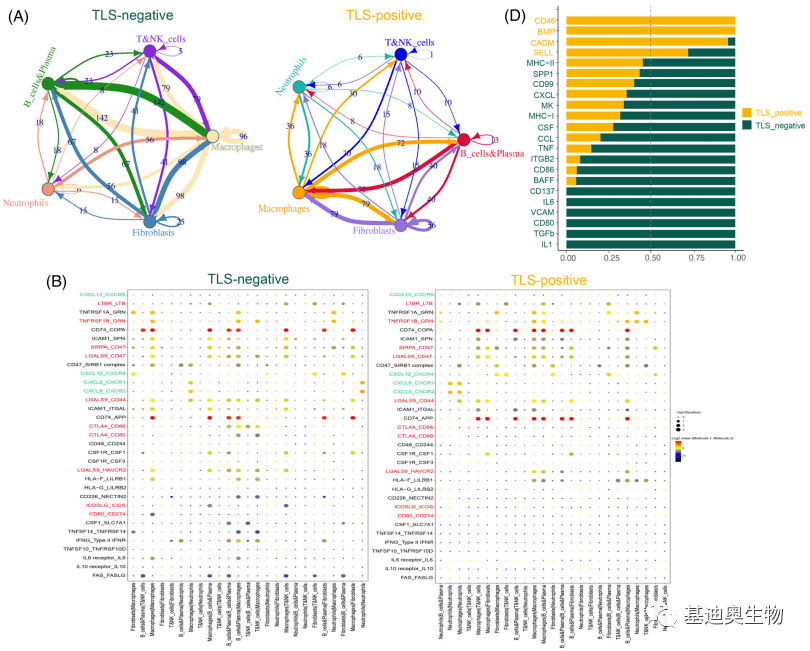
图3 肿瘤微环境的细胞通讯分析
4.scRNA-seq揭示了不同TLS状态下TAN的可塑性
最近的研究表明,TAN积极参与B细胞向TME的募集,TNFα是介导B细胞趋化的主要细胞因子,TAN上的膜B细胞活化因子(BAFF)是介导B细胞分化的潜在接触机制我们在研究中发现了一个有趣的现象:在TLS阴性组中,TAN的BAFF和TNF水平相对较高。由于我们的scRNA-seq数据集样本量较小,使用反卷积算法CIBERSORTx来模拟细胞类型特异性基因表达谱,并预测大规模scRNA-seq数据集中每种细胞类型的丰度。为了更好地了解BC微环境中TAN和TIL-B之间的关系,我们检查了BC队列中两种细胞类型及其亚群浸润模式的配对Spearman相关性,将它们分为两组:TLS-low和TLS-high。与TLS-high组相比,TLS-low组的TAN与TIL-B呈显著正相关。
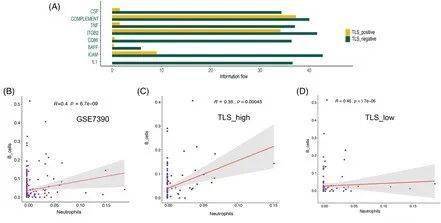
图4 与普通转录组关联的去卷积分析
研究结论
本研究强调了BC中的B细胞异质性,解释了B细胞和TLS如何在单细胞和临床水平上显著促进抗肿瘤免疫。
关于这两种转录组关联的应用还有多种,后期发现更好的文献再给大家分享。如果大家有该方面的项目意向,也可以联系基迪奥客服或当地销售,我们有丰富的项目经验。
参考文献
[1] Wang, Long et al. The maturation and aging trajectory of Marchantia polymorpha at single-cell resolution. Developmental Cell, VOLUME 58, ISSUE 15, P1429-1444.E6, AUGUST 07, 2023.
[2] Wang, Q, Sun, K, Liu, R, et al. Single-cell tranome sequencing of B-cell heterogeneity and tertiary lymphoid structure predicts breast cancer prognosis and neoadjuvant therapy efficacy. Clin Transl Med. 2023; 13:e1346.
*未经许可,不得以任何方式复制或抄袭本篇文章之部分或全部内容。版权所有,侵权必究。
基迪奥生物|专业定制测序服务
联系方式:020-39341079;service@genedenovo.com